多細胞生物の発生過程では、1つの受精卵が次々に分裂し、多様に異なるタイプの細胞へ分化することによって、複雑な体制を持つ個体が形成されます。この過程を司る遺伝子やタンパク質などの構成要素は次々に同定されていますが、一方で、化学反応レベルでのゆらぎや、しばしば変動する環境下において、どのようにこの発生過程が安定に進行するか、といった問題は不明な点が多く残されています。こうした問題は、細胞内外の構成要素を調べ上げるのみではなく、それらの絡み合いによって生じるシステムとしての性質として理解すべきものだと考えられます。
そこで私達は、内部に化学反応や遺伝子制御のネットワークを持つ細胞が相互作用している簡単な多細胞モデルを計算機上に構築して、そのシステムにおいて普遍的に見られる性質を調べています。ここで普遍的とは、モデルの細部に依存せずに出現することを意味しています。その結果、多様な状態に分化する能力を持つ細胞(幹細胞と呼ばれています)は、時間的に複雑に振動する内部ダイナミクスを持ち、そこから分化してきた細胞は、より単純な内部ダイナミクスを持つことが明らかになりました(下図を参照)。また、その複雑な内部ダイナミクスを幹細胞からの分化は、周囲に存在する細胞の構成比によって勝手に制御され、結果として細胞集団レベルでの安定性を持つことが示されました。
この細胞シミュレーションの結果は、ES細胞や造血幹細胞、あるいは受精卵といった分化能を持つ細胞においては、複雑に振動する遺伝子発現のダイナミクスが存在することを予言しています。実際に最近になって、ES細胞の内部状態が大きな多様性を持ち、一部の遺伝子が時間的に振動していることが実験的に見いだされています(例えば、Hayashi et al., Cell Stem Cell 2008,3:391-401やChambers et al., Nature 2007, 450:1230-1235を参照して下さい)。私達は、こうした細胞シミュレーションからの帰結と、幹細胞の1細胞レベルでの実験的解析を組み合わせることにより、多細胞生物のシステムとしての性質を理解することを目指しています。
この研究の詳しい情報は、以下の文献を参照して下さい。
A dynamical-systems view of stem cell biology, Chikara Furusawa, Kunihiko Kaneko, Science 338(6104):215-7 (2012)
Chaotic expression dynamics implies pluripotency: when theory and experiment meet, Chikara Furusawa, Kunihiko Kaneko, Biology Direct, vol.4:17(2009) PDF
Theory of robustness of irreversible differentiation in a stem cell system: chaos hypothesis, Chikara Furusawa, Kunihiko Kaneko, J. Theor. Biol. 209, 395-416 (2001) PDF
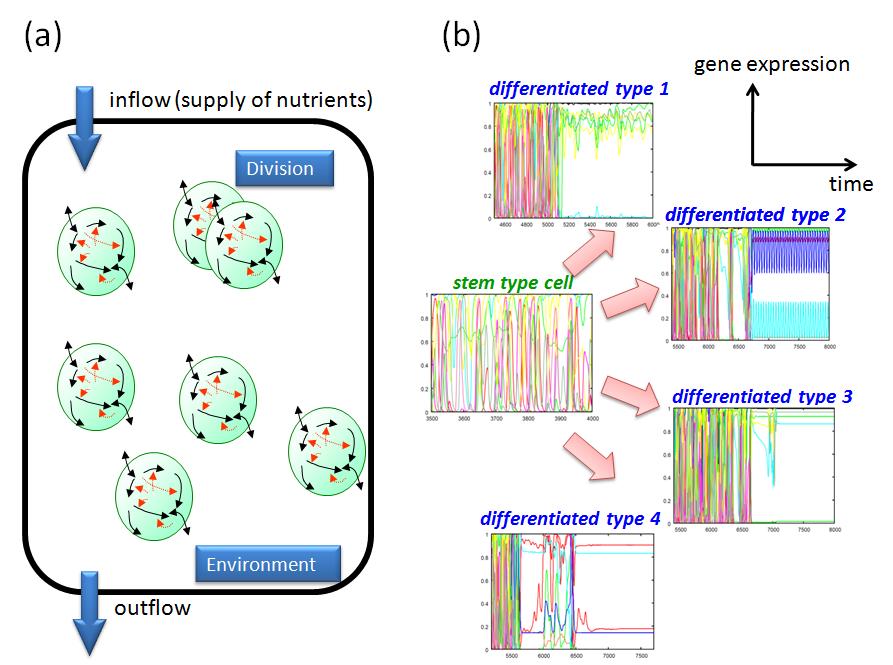
図(a): モデルの模式図。内部に遺伝子発現などの化学反応のネットワークを持つ細胞が、分裂をしつつ環境を通じて相互作用している。図(b):相互作用による細胞分化過程の一例。それぞれの図は、複数の遺伝子発現量のダイナミクスを示している。細胞数が小さい場合には、全ての細胞は図中の"stem type cell"とされた複雑に振動する細胞状態を持つが、分裂によって細胞数が増加することによって系全体が不安定となり、一部の細胞が別のより単純なダイナミクスを持つ状態へ遷移する("differentiated type"の状態)。
今日では多くの微生物のゲノムが解読され、細胞内の代謝反応のほとんどを同定することが可能となっています。そうしたゲノムスケールの代謝ネットワークの情報を基に、微生物の代謝の流れを予測することができれば、工学的な応用への代謝のデザインが可能となります。そこで我々は、ゲノムスケールで代謝の流れ(フラックス)を計算機上でシミュレートし、その結果と実際の実験結果の対応を解析しています。実験とシミュレーションの対応から、モデルや計算方法の改良を行い、予測精度の向上を目指しています。
我々の代謝のシミュレーションでは、細胞状態が定常であることを仮定し、線形代数を基盤とした代謝フラックスの解析を行っています。これは、酵素の反応定数などの現状では未知のパラメータを用いずに、細胞がとり得る状態を計算できるというメリットのためです。この定常状態の仮定と、適切な目的関数(例えば、細胞の増殖速度)を最適化する計算を行うことにより、細胞の代謝状態を予測出来ることがわかってきました。我々 は、物質生産において広く利用されるコリネ型細菌Corynebacterium glutamicum のゲノムスケール代謝モデルを構築し、そのシミュレーション結果が実験によって観測される代謝状態とよく対応することを見いだしています(下図を参照)。また、酵母や大腸菌などのゲノムスケール代謝モデルを用いて、遺伝子操作が代謝状態に与える影響をシミュレートし、その結果を実験的に確認しています。こうした代謝シミュレーションによる予測は、代謝を合理的にデザインする上で重要なツールになると期待されています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Development and Experimental Verification of a Genome-Scale Metabolic Model for Corynebacterium glutamicum, Yohei Shinfuku, Natee Sorpitiporn, Masahiro Sono, Chikara Furusawa, Takashi Hirasawa and Hiroshi Shimizu, Microbial Cell Factories, vol. 8, NO. 43(2009)
PDF
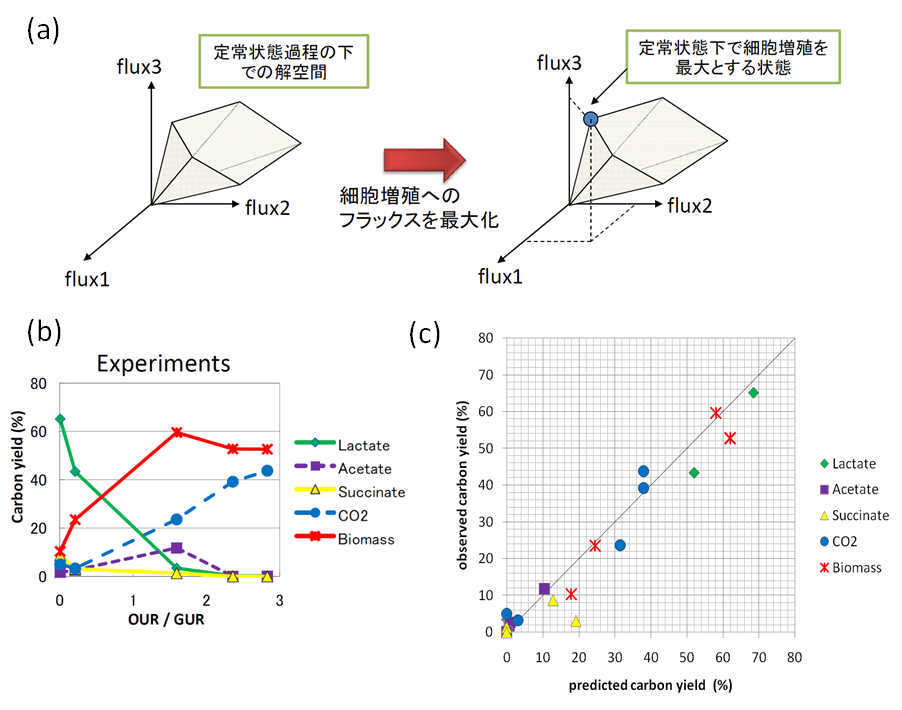
図(a):代謝状態が定常である(各代謝成分について生成フラックスと消費フラックスがバランスする)と仮定すると、 取り得る代謝状態を示す解空間を定義できる。この解空間内で、細胞増殖を表す仮想的な細胞合成フラックスを 線形計画法によって最大化 することにより、定常状態を満たしながら細胞増殖を最大とする代謝状態を計算できる。図(b):この手法の有効性を 検証するため、Corynebacterium glutamicum を5種類の異なる酸素取り入れ条件の下で培養し、そのときの 炭素収率をプロットしている。横軸はグルコース取り入れ量(GUR)で正規化した酸素取り入れ量(OUR)、縦軸は 細胞、二酸化炭素、有機酸の炭素収率を示す。図(c):シミュレーション結果の予測精度を示すため、横軸に実験結果と同じ GURとOURにおいて増殖速度を最大としたときの炭素収率、縦軸に対応する図(b)に示した実験結果をプロットしている。 図に見られるように、シミュレーションは高い予測精度を持つ。この手法を用いて、遺伝子操作や環境変動に応じて 代謝状態がどのように変動するかを予測することが可能となる。
生物システムは、その環境変化に応じて柔軟に内部状態を変化させ、その環境に適応する能力を持ちます。例えば、大腸菌のような微生物を(これまでに経験のしたことがないような)一定のストレス環境下で長期間(例えば数十日)培養すると、ゆるやかに増殖速度が上昇するといった適応進化の現象が見いだされます。これは、遺伝子ネットワークの応答反応のような表現型の変化と、ゲノム配列の変化、つまり遺伝子型の変化の双方が絡み合った現象である可能性が高いのですが、その詳細は理解されていません。こうした生物システムの柔軟な環境適応を解析することは、複雑な細胞システムがどのように適応・進化をすることが出来るかといった生物学の本質的な問題を扱うと同時に、生物を用いた物質生産過程における生産性向上や、生物的な柔軟性・安定性を持つ人工システムのデザインなど、工学的な応用においても重要な意味を持ちます。
そこで私達は、大腸菌を用いて、エタノールストレス環境下での長期間の植え継ぎ培養を行い、その過程における表現型と遺伝子型の網羅的解析を行っています。表現型の変化については、高密度タイリングアレイを用いて高精度の遺伝子発現解析を行い、遺伝子型の変化についても、同じタイリングアレイを用いた網羅的なゲノム変異解析を行っています。また、同一環境での複数の植え継ぎ培養を独立に行うことにより、その過程のおいて共通に生じる変化を抽出することを試みています。結果として、およそ2500時間の培養により、独立に培養した全ての系列で比増殖速度が1.5倍程度上昇することが見いだされています(下図を参照)。また、遺伝子発現解析の結果から、それらの系列において共通に生じた表現型変化を抽出することが出来ています。さらに、網羅的なゲノム配列変化の情報を取得することによって、表現型と遺伝子型の変化の対応を議論することを試みています。こうした長期間培養による人工進化実験をさまざまな環境下で行うことによって、生物システムの環境適応の理解を目指しています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Transcriptome analysis of parallel-evolved Escherichia coli strains under ethanol stress, Horinouchi T. et al., BMC Genomcics, vol.11:579 (2010) PDF
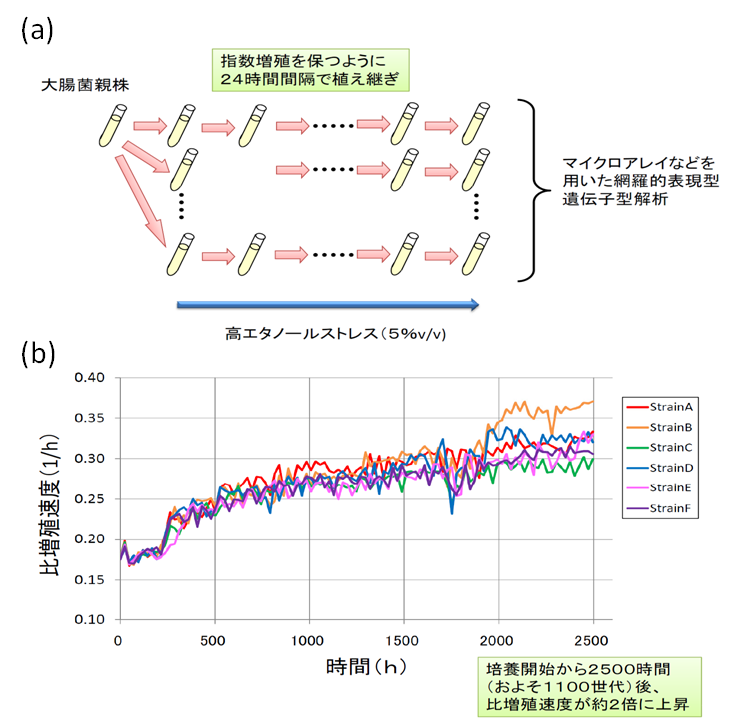
図(a):植え継ぎ実験の概念図。試験管を用いた24時間間隔の植え継ぎ実験を長期間にわたって行うことにより、 その環境において適応進化した大腸菌株を取得する。そして、その適応進化の過程において、表現型/遺伝子型に どのような変化が生じたのかを網羅的に測定することにより、その耐性出現のメカニズムを理解し、 それに基づいた工学的に有用な微生物の育種を目指す。図(b):長期植え継ぎ培養結果の一例。この例では5%エタノール ストレス環境下において6つの 独立系列の植え継ぎ培養を約2500時間行ったところ、親株と比較して比増殖速度が1.5倍程度上昇した株を取得することに 成功している。
Affymetrix社のGene-chipシステムに代表されるDNAマイクロアレイでは、基盤上で合成した数百万種類といった膨大な数のDNAプローブ分子に対して、蛍光標識したDNA又はRNAサンプルをアプライすることにより、それらの間の二本鎖形成を利用して、サンプル中の特定ターゲット配列の濃度を定量することが可能です。この実験系を用いることにより、細胞内の全遺伝子発現量の網羅的な定量や、ゲノム配列の変異によるシグナル低下を利用したゲノム変異解析が可能となっています。しかしながら、膨大な種類のDNA/RNAターゲットを同時にアプライするという実験方法においては、非特異的な二本鎖形成によるノイズの発生や、二本鎖形成時の非線形的な振る舞いを避けることが出来ないため、従来の解析アルゴリズムでは必ずしも高い測定精度や広い測定レンジを持つことが出来ませんでした。
そこで私達は、基盤上のプローブDNAとサンプル中のターゲットDNA/RNAとの二本鎖形成の物理モデルを構築し、それを用いたマイクロアレイの高精度解析アルゴリズムを開発しました。そこでは個々のプローブDNAの配列から、そこで特異的又は非特異的に二本鎖形成が起こる時の結合エネルギーを計算し、二本鎖形成の物理モデルを利用してターゲットの定量を行います。そのアルゴリズムを用いることにより、遺伝子発現量の測定レンジを従来手法と比較して2桁程度拡大することに成功しました(下図を参照)。さらに、蛍光標識したゲノムDNAをアプライすることによるゲノム変異解析についても、同様の物理モデルを利用することによって高い精度での1塩基置換の同定を可能としています。こうした解析アルゴリズムを用いることによって、細胞の表現型と遺伝子型を高精度で網羅的に測定することが可能となり、それらの関係を理解することによってシステムの適応・進化のメカニズムの理解や、その情報に基づく工学的に有用な微生物の育種を目指しています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Model-based analysis of non-specific binding for background correction of high-density oligonucleotide microarrays, Chikara Furusawa, Naoaki Ono, Shingo Suzuki, Tomoharu Agata, Hiroshi Shimizu, Tetsuya Yomo, Bioinfromatics, Vol.25, No.1, p.36-41(2009)
PDF
An improved physico-chemical model of hybridization on high-density oligonucleotide microarrays, Naoaki Ono, Shingo Suzuki, Chikara Furusawa, Tomoharu Agata, Akiko Kashiwagi, Hiroshi Shimizu, Tetsuya Yomo, Bioinformatics, Vol.24, No.10, p.1278-1285 (2008)
PDF
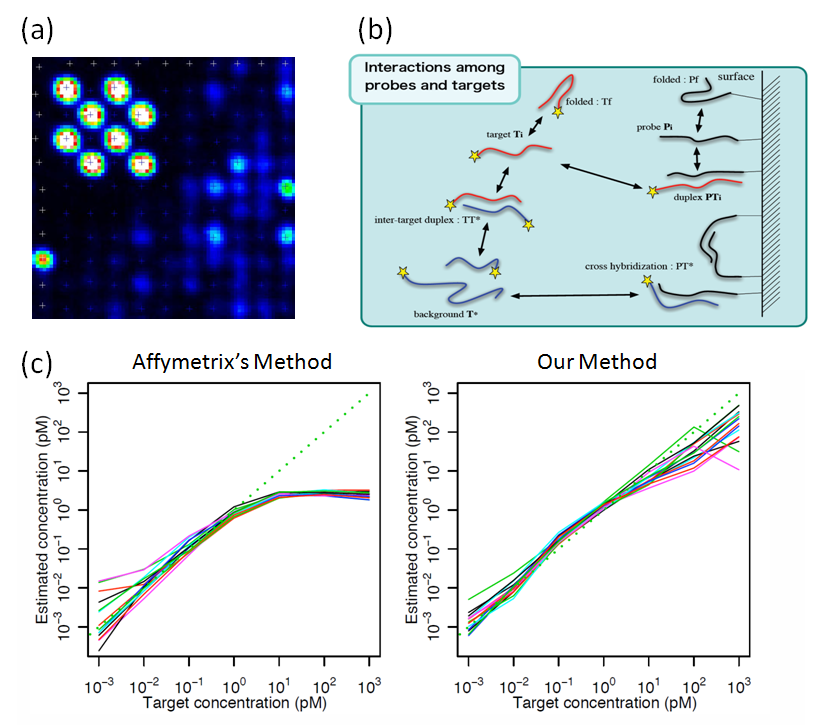
図(a):マイクロアレイ(Affymetrix社 gene-chipシステム)の画像の例。各蛍光スポットは異なる配列を持つ DNAプローブが存在している領域に対応しており、その蛍光強度から、プローブに相補的な配列を持つターゲットの濃度を定量する。図(b):本研究で構築したハイブリダイゼーション反応の物理モデルの概念図。 プローブとターゲットのハイブリダイゼーションの他、非特異的な反応や、二次構造の形成を考慮に入れている。 ハイブリダイゼーションの自由エネルギーをNearest Neighbor法を用いて推定し、平衡反応を仮定して 蛍光強度を予測するモデル式を構築している。図(c):ターゲット濃度の予測精度の検証。この実験では、既知濃度のmRNAをサンプルに導入してアレイ実験を行い、その濃度の定量を2つの異なる手法で行っている。ぞれぞれの図において、横軸に投入したmRNAの濃度を、縦軸に推定されたmRNA濃度をプロットしている。左図のAffymetrix社が提供する手法(MAS5.0)と比較して、図(b)で示す物理モデルを用いた手法では、2桁程度のダイナミックレンジの拡張に成功している。
細胞は、環境変化に応答するための様々な制御ネットワークを持っています。例えば、ある栄養素が枯渇した場合には、細胞内の遺伝子発現プロファイルが変化して、別の栄養素が利用できる状態に変化します。こうした応答機構は計算機プログラムにおけるIf-Then型の制御のように、その入力と出力の関係が決まっています。しかし、細胞の柔軟な環境適応は、こうしたIf-Then型の応答で全て記述できるのでしょうか?最近の柏木らによる大腸菌内に人工の遺伝子制御ネットワークを導入した研究では、大腸菌はこれまでに経験したことがない環境変化に対しても、適切に応答できることが示されています(Kashiwagi et al., PLoS One 1(1): e49)。また、この環境変化に対する応答のモデルとして、遺伝子発現量のゆらぎが重要であるという「アトラクター選択」の考え方が提唱されています。そこでは、細胞の状態に応じて実効的なゆらぎの大きさが変化することにより、細胞にとって不利な状態(アトラクター)からはゆらぎによってすぐに脱出し、有利な状態では長く留まる可能性が示唆されています。
そこで私達は、この考えをより大規模な遺伝子-代謝ネットワークに拡張したモデルを構築し、細胞が複雑な環境変化に対しても応答が可能であるかを検証しました。その結果、十分に大きい数のアトラクターを持つ遺伝子制御ネットワークを使うことによって、細胞は任意の環境変化に対して柔軟に内部状態を変化させ、その環境下で増殖可能な状態を選択できることが示されました。また、こうしたゆらぎを利用した適応過程が存在することによって初めて、実際の細胞内に存在する精巧な遺伝子制御ネットワークが出現したことが示唆されています。こうした結果は、文部科学省グローバルCOEプログラム「アンビエント情報社会基盤創成拠点」や、文部科学省振興調整費「生体ゆらぎに学ぶ知的人工物と情報システム」などのプロジェクトを通じて、ロボットやインターネットなど様々なシステムのデザインに役立てるための研究が進んでいます。
この研究の詳しい情報は、以下の文献を参照して下さい。
A generic mechanism for adaptive growth rate regulation, Furusawa, C., and Kaneko, K., PLoS Computational Biology, Vol.4(1), p.e3 (2008, Jan)
PDF
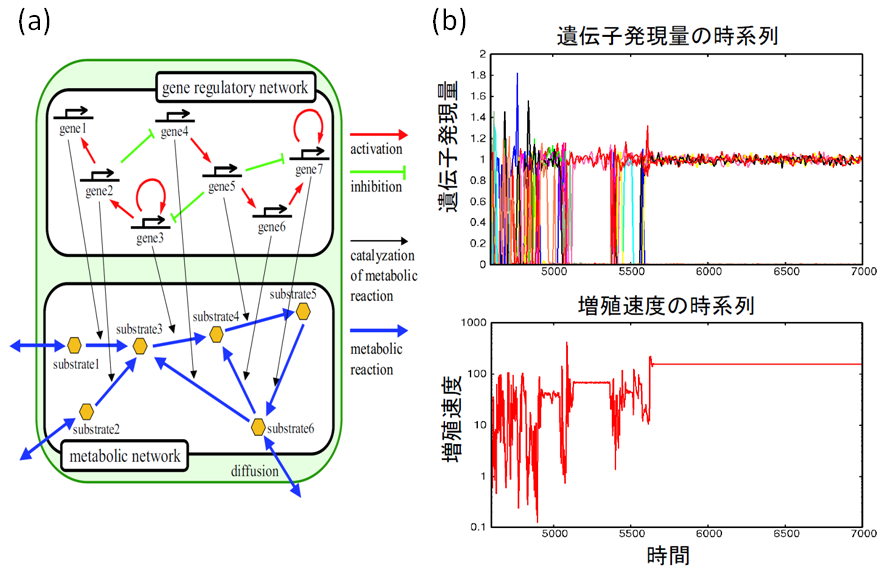
図(a):遺伝子-代謝ネットワークを持つ細胞モデルの概念図。このモデルは以下の性質を持つ。1)多数の遺伝子が互いに発現を促進/抑制し合い、結果として多数の安定な状態が存在する。2)遺伝子は代謝反応を触媒し、その代謝反応の結果として細胞の増殖速度が決まる。3)遺伝子の発現量と分解量はそれぞれ細胞の増殖速度に比例する。 4)遺伝子発現量には、分子の小数性に起因する時間的なゆらぎが存在する。図(b):シミュレーション結果の一例。この系では、細胞の増殖速度が小さい場合には、遺伝子のダイナミクスに対してゆらぎの効果が相対的に大きくなり、別の状態への遷移を誘起するのに対し、増殖速度が大きい状態では、その効果が小さくなる。図の時系列において、増殖速度が小さい前半では、遺伝子発現のゆらぎが大きい。一方で、時系列の後半に見られるように、一度増殖速度が大きい状態に遷移すると、実効的なゆらぎが小さくなることによってその状態から脱出することが出来ない。結果として、複雑な制御機構を持たずとも、高い増殖速度を持つ状態をゆらぎによって選択することが可能となる。
マイクロアレイなどの網羅的な解析技術の発展は、細胞内の化学反応ネットワークについての膨大な情報をもたらしています。こうした膨大な情報は、現在の生物学の主流である生物システムの分子レベルでの記述とは異なる、分子の詳細に依存しないマクロレベルでの記述の可能性をもたらしています。ここでマクロレベルの記述とは、下のレベルにおける情報を粗視化したレベルでの記述のことで、例えばコップの中の水の状態を記述するときに、水分子の一つ一つを詳細に記述することも可能ですし、より粗視化した記述として、その水の温度やエントロピーといった個々の分子の詳細に依存しない統計的な量を用いることも出来ます。物理学においては、こうした異なるレベルでの記述とそこでの法則、そして異なる階層間の関係を明らかにすることが重要とされてきました(例えば熱力学と統計力学)。一方で生物学においては、遺伝子やタンパク質などの分子レベルでの記述に重きが置かれ、それ以外の階層での記述は扱われてきませんでした。しかしながら、生物のシステムとしての安定性や不可逆性を理解する上で、分子の詳細に依存しないマクロレベルでの記述が有効である可能性も考えられます。
そこで私達は、マイクロアレイやSAGE (Serial Analysis of Gene Expression) による網羅的発現解析のデータや、フローサイトメトリによる1細胞状態測定データを用いて、分子の詳細に依存しないマクロレベルでの統計的な性質について解析をしています。その一つの結果として、細胞内の各遺伝子の発現量の多い順に並べてみて、量と順位の関係をみてみると、その発現量と順位に逆比例するという法則が見いだしています(下図を参照)。この性質は、生物種や細胞の種類に依存せずに常に出現するものであり、細胞内ダイナミクスが持つ普遍的な性質であると言えます。また、同一遺伝子型・同一環境にある細胞集団について、ある遺伝子の発現量を1細胞レベルで測定したところ、それらの発現量の分布は対数世紀分布に従うことが見いだされています。このゆらぎの性質も、遺伝子や細胞の種類に依存しないことが示唆され、細胞内ダイナミクスが持つ普遍統計則であると考えられます。こうした統計則の起源を理解するために、外部から栄養と取り込みながら分裂する細胞モデルのシミュレーションを行いました。その結果として、モデルの細部に依存することなく、細胞が同じ状態を自己複製することが出来る臨界状態において、上記の2つの統計則が常に出現することが確認されました(下図を参照)。こうした細胞内ダイナミクスが従う(個々の分子レベルに依存しない)統計則を解析することにより、細胞システムが持つ性質をマクロレベルで記述する論理体系の構築を目指しています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Zipf's Law in Gene Expression, Furusawa, C., and Kaneko, K.,
Physical Review Letters, 90(8), 088102 (2003)
PDF
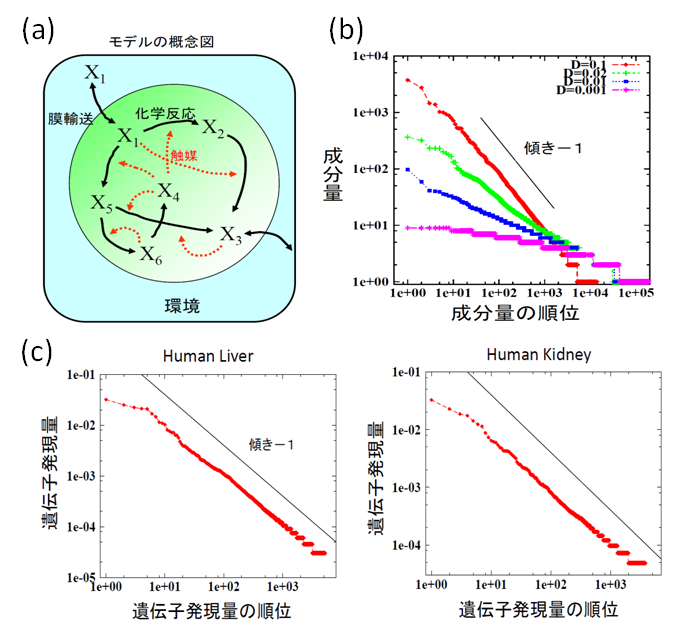
図(a):モデルの概念図。内部に複数の化学成分X1, X2,…, Xnが存在し、それらが触媒反応のネットワークを持つとしている。また、一部の成分は膜を拡散によって透過することが可能であり、そのときの拡散係数をDとする。環境から栄養となる成分を取り込み、それを化学反応によって別の成分に変換する結果として、細胞内の成分の総量は増加する。その成分の総量を体積に相当するものと考え、それが一定値を超えたら半分の成分からなる細胞へと分裂するとしている。図(b):拡散係数Dを変化させて、細胞内の成分量のランク分布(横軸を成分量の順位、縦軸を成分量)をプロットしている。細胞の分裂速度を最大とするDc(図ではDc〜0.1)において、成分量のランク分布は傾き−1のベキ則(Power-Law)に従う。この性質はモデルの細部に依存しない、分裂していく細胞系における普遍的性質である。図(c):実験的に得られた細胞内の遺伝子発現量のランク分布。左は肝細胞、右は腎細胞の遺伝子発現量を、SAGEによって解析したデータのプロットとなっている。図に見られるように、遺伝子発現量のランク分布はほぼ傾き−1のベキ則にしたがう。この性質は、細胞タイプや生物種に依存せずに広くみられる性質であり、細胞内の反応ダイナミクスが従う普遍則であることを示唆している。
酵母や大腸菌など、有用物質の生産する能力や産業上有用な特性を持つ微生物を遺伝子操作により改良することは、それらを工学的に応用する上で重要な意味を持ちます。我々は、DNAマイクロアレイと呼ばれるツールを使うことにより、細胞内の遺伝子発現量を網羅的に測定し、その情報から微生物の改良に有用な遺伝子の探索を試みています。
そうした研究の一例として、我々は酵母のエタノール耐性について解析しています。エタノールは酵母の主要な生成物の一つですが、その生産過程では、高濃度のエタノールによるストレスが酵母自身に影響を与え、生産性の低下をもたらします。そこで、エタノールストレスへの耐性の異なる2つの酵母株の 遺伝子発現プロファイルを比較することにより、その耐性メカニズムに関与する遺伝子の抽出を試みました。DNAマイクロアレイで得られる網羅的な遺伝子発現データは膨大な情報量を持つので、クラスタリング解析、ネットワーク解析や統計解析など、情報科学からの知見を応用して解析を行いました。その結果、酵母のエタノール耐性にはアミノ酸であるトリプトファンの合成が関与することを見いだし、実際にトリプトファン合成能を強化した酵母株を作成したところ、エ タノール耐性能を示しました。こうした網羅的解析を用いた手法は、ストレス耐性のみならず、生分解性ポリマーの原料など、有用物質を生産する微生物の生産性向上にも適応されています
この研究の詳しい情報は、以下の文献を参照して下さい。
Identification of target genes for conferring ethanol stress-tolerance to yeast Saccharomyces cerevisiae based on DNA microarray data analysis, Hirasawa, T., Yoshikawa, K., Nakakura, Y., Nagahisa, K., Furusawa, C., Katakura, Y., Shimizu, H. and Shioya, S., Journal of Biotechnology, Vol.131, p.33-44 (2007)
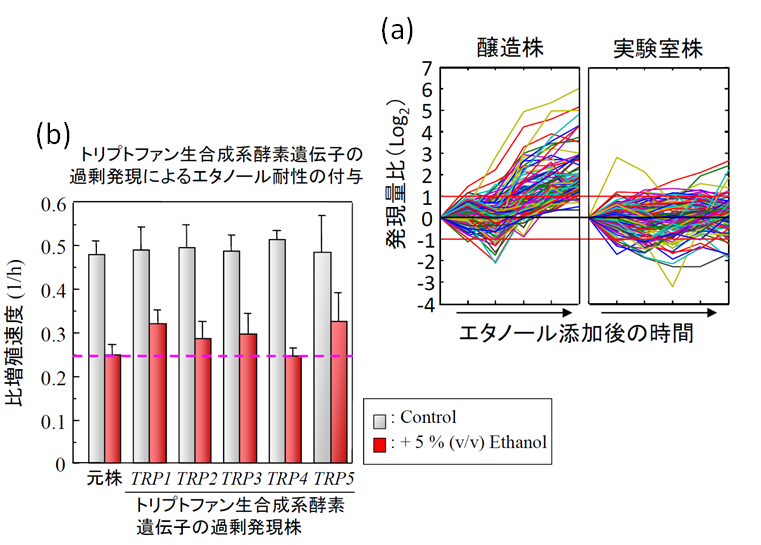
図(a):酵母のDNAマイクロアレイを用いて、エタノール耐性を持つ醸造株(工業的に用いられる株)と、実験室株におけるエタノールストレス添加後の遺伝子発現量変化を網羅的に定量した。クラスタリング解析の結果、醸造株において特異的に発現量が上昇する遺伝子群が同定され、解析の結果そのクラスタにはトリプトファンなどのアミノ酸合成に関与する遺伝子が有意に多く含まれていた。図(b):マイクロアレイ解析の結果から、トリプトファン合成経路のエタノール耐性への関与の可能性が示唆された。そこで、トリプトファン合成経路上の遺伝子発現を増強した実験室株を遺伝子操作によって作成したところ、エタノールストレス環境下で操作前の元株に対して有意に増殖速度の高い株を得ることに成功した。
我々は、DNAマイクロアレイなどの網羅的遺伝子発現情報から、操作を加える候補遺伝子を探索し、遺伝子操作によって工学的に有用な特定の賦与を目指しています。しかしながら、どのように網羅的情報から候補遺伝子を探索するか、また遺伝子操作の影響がどのように細胞の表現型に影響を与えるかについて、未だ理解が進んでいない部分が残っています。これは、遺伝子の発現とそれに付随する細胞の表現型との間にどのような関係性があるのかを研究者自身がきちんと把握できていないからであると考えられます。もし遺伝子発現の網羅的な情報から細胞の表現型に迫ることが可能であるならば、遺伝子発現情報から目的とする育種の結果を予測することができるようになると期待され、効率的な生物創製の技術へとつながると期待されます。
そこで私たちは、4,000株以上ある酵母の1遺伝子破壊株や1遺伝子発現増強株を用いて、ストレスに対する増殖挙動 (表現型)を網羅的に解析しました。このデータと網羅的な遺伝子発現情報から、遺伝子破壊という摂動に対する表現型の変化と、そのときの遺伝子発現量変化 の関係を明らかにしようと試みています。そのために、ハイスループットかつ実験の再現性の高い96穴マイクロタイタープレートを用いた培養系を構築しました。その結果、ペルオキシソームの形成に関与する遺伝子がエタノールストレスと関係があるという新しい知見を得ました。また、ストレス環境下で発現が低下する遺伝子は、破壊された場合に高い確率で増殖速度の低下を引き起こし、発現が増加する遺伝子の破壊はむしろ増殖が変化しない確率が高いことがわかってきました。このような解析結果がストレス耐性を示す細胞を創製する際に有効であるのか検討しています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Comprehensive phenotypic analysis for identification of genes affecting growth under ethanol stress in Saccharomyces cerevisiae, Katsunori Yoshikawa, Tadamasa Tanaka, Chikara Furusawa, Keisuke Nagahisa, Takashi Hirasawa, Hiroshi Shimizu, FEMS Yeast Research, Vol.9, Issue1, p.32-44(2009)
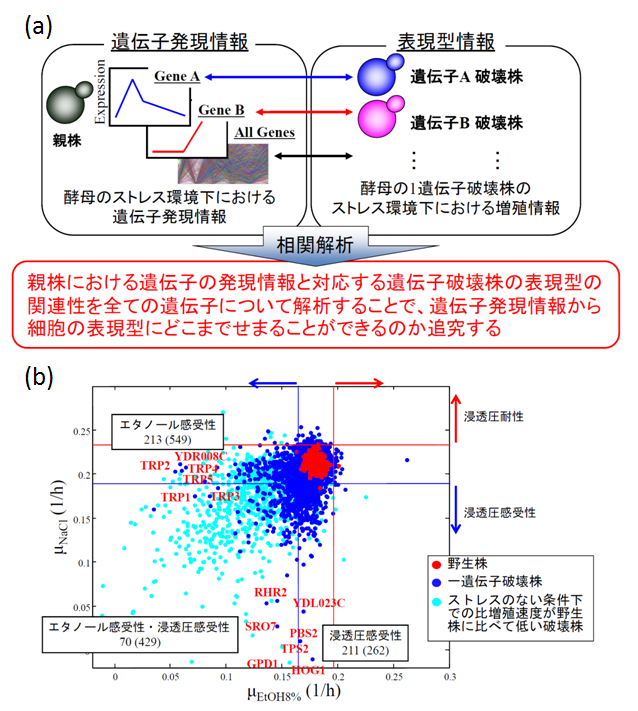
図(a):研究の概念図。網羅的に遺伝子発現量を定量することができるマイクロアレイによる解析と、4000株程度ある酵母の1遺伝子破壊ライブラリによる網羅的表現型解析を組み合わせることにより、遺伝子発現のダイナミクス(表現型)と1遺伝子破壊の表現型への影響(遺伝子型)との相関を解析する。図(b):網羅的表現型解析結果の1例。この実験では、96穴プレートによる培養系を用いて、1遺伝子破壊による比増殖速度の変化を2つのストレス環境下(NaClによる浸透圧ストレス、エタノールストレス)において測定している。図の赤、青のそれぞれの点は、親株、1遺伝子破壊株の増殖速度の結果に対応しており、水色の点は非ストレス環境下において増殖速度が親株に比べて有意に低い株を表している。この実験結果から、破壊することによってエタノール感受性や浸透圧感受性になる遺伝子をゲノム上のほぼ全ての遺伝子中からスクリーニングすることが可能となる。
近年の実験技術の向上により、細胞内遺伝子発現ダイナミクスの高精度の測定が可能となっています。その結果として、同一の遺伝子型で均一な環境に ある微生物集団においても、その内部状態には大きな「揺らぎ」が存在することが解ってきました。この揺らぎは、細胞内の化学反応の確率的な振る舞いに起因すると考えられています。こうした揺らぎの存在は、生物というシステムの本質に迫る上で、重要な役割を持つと考えられています。例えば、生物は一般に、環境が変化しても柔軟に内部状態を変化させ、その環境に適応することが可能ですが、そうした柔軟さを理解する上でも揺らぎの存在とその性質を理解することが重要であると予想されています。
そこで我々は、フローサイトメトリーと呼ばれる個々の細胞レベルでの細胞状態の測定を可能とする機器を用い、GFP蛍光タンパクを利用して細胞の遺伝子発現量の揺らぎを測定し、その性質の理解を目指しています。例えば、大腸菌の連続培養系を用いて、細胞の増殖速度と揺らぎの関係を解析しました。その結果、細胞の増殖速度がある程度低くなると、突然細胞内ダイナミクスにおける揺らぎが大きくなる現象が見いだされました。こうした知見は、細胞内ダイナミクスにおける揺らぎの性質の理解の手がかりになると共に、安定なバイオプロセスのデザインにも応用が可能であると考えられます。
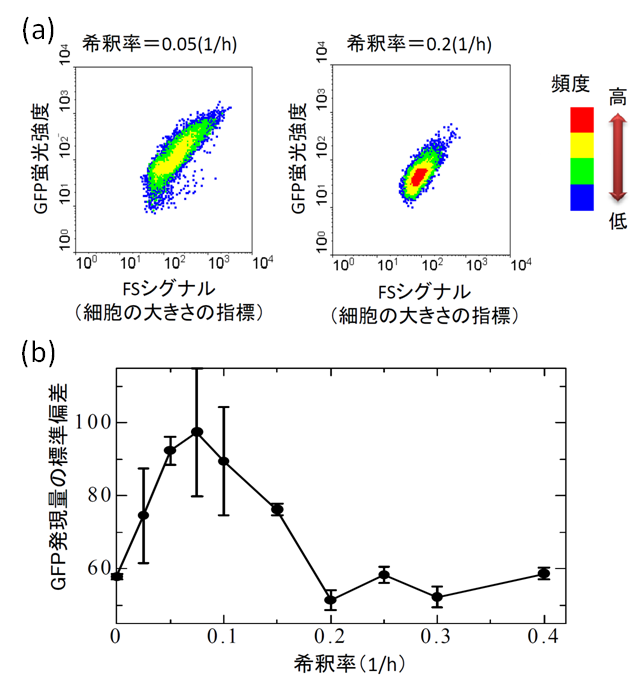
図(a):大腸菌の連続培養系における細胞状態のフローサイトメトリーによる解析。横軸は前方散乱光(Forward Scatter Signal)、縦軸はGFP蛍光強度を示す。ここで、GFP蛍光強度は細胞内のタンパク質合成活性の指標として用いている。定量は1細胞レベルで行い、その密度分布図をプロットしている。希釈率が比較的高い場合(右図)では、細胞の状態は比較的一様であるが、希釈率が低い(=細胞の増殖速度が小さい)場合には、細胞状態は相対的に多様となり、つまり状態が大きく揺らぐ。また、この希釈率が小さい状態においてのみ、分布が時間的に変動する現象が観測されている。図(b):GFP蛍光シグナルの標準偏差の希釈率への依存性。図(a)で示したように、細胞状態の多様性は希釈率に依存して変化する。そこで、その状態多様性の指標として、GFP蛍光シグナルの標準偏差(図(a)の縦軸の広がり具合の指標)を用い、それが希釈率によってどのように変化するかをプロットしている。図で見られるように、希釈率が0.15(1/h)より小さくなると、細胞状態の多様性が上昇する。
物質生産の効率化へ向けた代謝デザインを行うためには、遺伝子操作などによる細胞内の代謝の変化を高精度で定量する必要があります。そこで我々は、 安定炭素同位体(13C)を用いて、細胞内の代謝の流れ(フラックス)を解析しています。この解析手法では、 13Cを含む炭素源(例えばグルコース)で微生物を培養し、その菌体を回収して、細胞内の代謝物質のそれぞれに13Cがどのように濃縮されているかを、質量分析機(GC-MSやCE-MS)や核磁気共鳴(NMR)を用いて解析します。その13Cの濃縮度のデータを基に、細胞内の代謝の流れを計算機上で再構成します。
例えば、この方法を用いて我々はコリネ型細菌Corynebacterium glutamicumのグルタ ミン酸生産期における代謝フラックスの解析を行いました。この研究では、細胞内のアミノ酸に含まれる13Cをガスクロマトグラフ質量分析器(GC-MS)を用いて解析しています。その結果、グルタミン酸を生産する時に活性化される代謝経路の同定に成功しました。こうした情報は、生産性のさらなる向上のため の遺伝子操作について、その指針を与えると期待されます。また、大腸菌や酵母の代謝フラックス解析も行っており、遺伝子操作や培養条件の変化による代謝フラックスの変化を定量することが可能となっています。こうした解析は、DNAマイクロアレイなどによる網羅的な細胞状態解析や、ゲノムスケール代謝モデルによる代謝予測と組み合わせることにより、代謝の合理的デザインを可能とするツールとして期待されています。
この研究の詳しい情報は、以下の文献を参照して下さい。
Study on roles of anaplerotic pathways in glutamate overproduction of Corynebacterium glutamicum by metabolic flux analysis, Shirai, T., Fujimura, K., Furusawa, C., Nagahisa, K., Shioya, S. and Shimizu, H., Microbial Cell Factories, Vol.6, No.1, p.19 (2007)
PDF
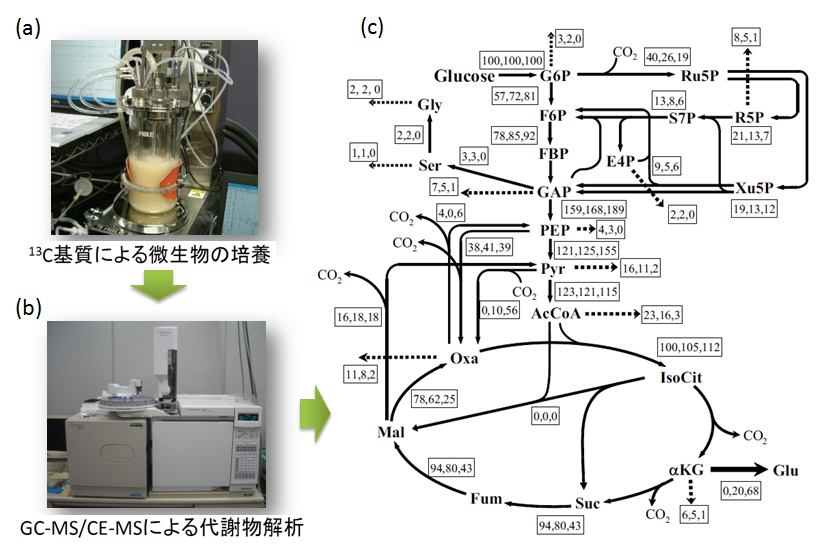
図(a):ジャーファーメンターによる微生物の培養。環境内の温度、pH、DO(溶存酸素濃度)などをコンピュータによってモニターし、制御を加えることが可能となっている。図(b):ガスクロマトグラフ質量分析器(GC-MS)。細胞内のアミノ酸やその他の代謝物における13Cの濃縮度を解析することが可能となる。この情報を基にして、コンピュータによって最適化計算を解き、得られた13C濃縮度を説明することが可能な代謝フラックスを推定する。図(c):フラックス解析結果の一例。この例では、コリネ型細菌Corynebacterium glutamicumのグルタ ミン酸生産期における代謝フラックスの解析を行っている。図中の枠内の数字は、左から細胞増殖期、グルタミン酸生産期(生産量小)、グルタミン酸生産期(生産量大)の、グルコース取り込みフラックスを100としたときの各代謝経路の代謝フラックスを示している。この解析結果から、グルタミン酸の生産期においては、ピルビン酸(Pry)からオキサロ酢酸(Oxa)への代謝フラックスの増加が生じていることが解る。
